В прошлом платформы данных о биоразнообразии строились на находках видового уровня, фактах обнаружения организма в пространстве и времени. С развитием молекулярно-генетических методов в изучении биоразнообразия, особенно методов метабаркодинга сообществ, большую роль играют данные молекулярного уровня.
Единицей информации в этом случае является факт обнаружения сиквенса в пространстве и времени (в результате выделения и секвенирования определенного участка ДНК). Такая информация должна быть опубликована, интегрироваться и быть доступной через платформы данных о биоразнообразии с тем же успехом, как данные видового уровня.
Другие страницы о молекулярных данных на сайте GBIF:
1) Подробная инструкция о публикации молекулярных данных на платформе GBIF (Publishing DNA-derived data)
2) Инструмент поиска по последовательностям GBIF Sequence ID tool
3) Включение видовых гипотез в систему GBIF (Adding sequence-based identifiers to backbone taxonomy)
4) Страница о ресурсах, доступных на портале GBIF по ДНК (GBIF and DNA)
5) Программа публикации данных метабаркодигна, в том числе инструмент для публикации таких данных (Metabarcoding data program)
6) Статьи с использованием сиквенсов на платформе GBIF (Featured data uses in biodiversity and DNA)
Молекулярные данные, полученные в результате изучения биоразнообразия, бывают следующих типов: 1) сиквенсы тотальной ДНК, выделенной в результате метабаркодинга субстратов, 2) сиквенсы ДНК, полученные в результате баркодинга (секвенирования) ваучерных образцов, 3) детекция целевых таксонов с помощью qPCR, 4) чеклист операционных таксономических единиц и связанных с ними таксонов, 5) просто метаданные.
| Данные мета(баркодинга) или количественной ПЦР(qPCR)? | ||||
| Мета (баркодинг) ↓ | qPCR ↓ | |||
| Молекулярная последовательность содержит географическую привязку в метаданных? | ||||
| Да ↓ | Нет ↓ | |||
| Молекулярная последовательность не имеет связанный с ней образов коллекции? | Набор данных имеет список видов таксонов и/или видовых гипотез? | |||
| Да ↓ | Нет ↓ | Да ↓ | Нет ↓ | ↓ |
| Категория I Находки ДНК в среде (тотальная ДНК) | Категория II ДНК образцов коллекций | Категория IV Списки видовых гипотез и таксонов | Категория V Только метаданные | Категория III Детекция целевых таксонов |
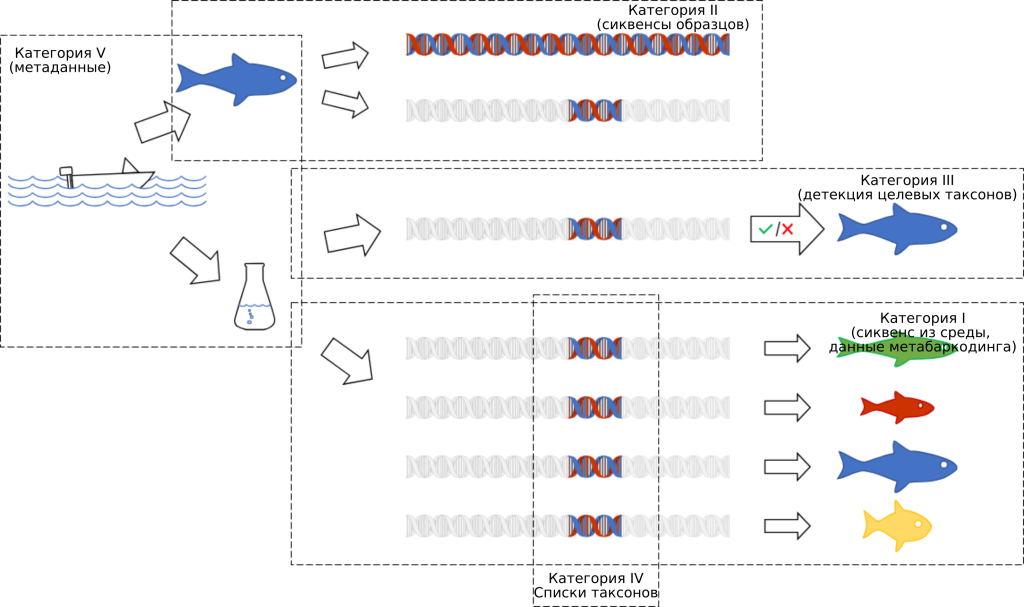
Примеры публикации сиквенсов на портале GBIF в виде наборов данных разных категорий:
1) Frøslev T, Ejrnæs R (2018). BIOWIDE eDNA Fungi dataset. Danish Biodiversity Information Facility. Occurrence dataset https://doi.org/10.15468/nesbvx accessed via GBIF.org on 2019-04-17.
2) PlutoF (2019). UNITE — Unified system for the DNA based fungal species linked to the classification. Version 1.2. Checklist dataset https://doi.org/10.15156/bio/587474 accessed via GBIF.org on 2019-04-17.
3) Cox F, Newsham K, Robinson C, Sweetlove M (2019). Microbial Fungi in soils on different Sub-Antarctic islands. SCAR — Microbial Antarctic Resource System. Metadata dataset https://doi.org/10.15468/jekfdj accessed via GBIF.org on 2019-04-17.
4) MGnify (2017). EMOSE (2017) Inter-Comparison of Marine Plankton Metagenome Analysis Methods. Sampling event dataset https://doi.org/10.15468/re7eoi accessed via GBIF.org on 2019-04-17.
5) Telfer A (2018). Centre for Biodiversity Genomics — Canadian Specimens. University of Guelph. Occurrence dataset https://doi.org/10.15468/mbwnw9 accessed via GBIF.org on 2019-04-17.
Для публикации данных сиквенсов используется специальное расширение IPT (DNA-derived data). Для публикации данных метабаркодинга используется две таблицы: таблица находок (occurrenceCore) и связанная с ней таблица сиквенсов (Dna-derived data). В таблице находок рекомендуется использовать специальный набор полей для описания основания находки, числа полученных ридов, уникальных номеров видовых гипотез, и другой специфичной для такого рода данных информации. Подробнее инструкция по публикации молекулярных данных на сайте GBIF (Publishing DNA-derived data through biodiversity data platforms).
Рекомендуемые поля данных и метаданных метабаркодинга (Occurrence core)
Название поля | Пример | Описание | Обязательное поле |
|---|---|---|---|
| basisOfRecord | MaterialSample | Основание находки (для молекулярных данных используется «MaterialSamples») | Обязательно |
| occurrenceID | urn:catalog:UWBM:Bird:89776 | Уникальный идентификатор наблюдения (молекулярной последовательности) | Обязательно |
| eventID | urn:uuid:a964765b-22c4-439a-jkgt-2 | Уникальный идентификатор события сбора (номер пробы, площадки, и т.д.) | Строго рекомендуется |
| eventDate | 2020-01-05 | Дата сбора образца | Обязательно |
| recordedBy | «Oliver P. Pearson | Anita K. Pearson» | Список людей — сборщиков материала или образцов с разделителем (‘ | ‘). | Строго рекомендуется |
| organismQuantity | 33 | Число ридов определеной видовой гипотезы (OTU или ASV) в образце. | Строго рекомендуется |
| organismQuantityType | DNA sequence reads | Размерность (число ридов ДНК) “DNA sequence reads”. | Строго рекомендуется |
| sampleSizeValue | 1233890 | Общее число ридов в образце (важно для пересчета числа ридов определенного таксона в процент относительно других таксонов в этой пробе). | Строго рекомендуется |
| sampleSizeUnit | DNA sequence reads | Размерность пробы (число ридов ДНК) “DNA sequence reads” | Строго рекомендуется |
| materialSampleID | https://www.ncbi.nlm.nih.gov/biosample/15224856 https://www.ebi.ac.uk/ena/browser/view/SAMEA3724543 urn:uuid:a964805b-33c2-439a-beaa-6379ebbfcd03 | Идентификатор materialSampleID (в отличие от конкретной цифровой записи материала образца). Используйте идентификатор биообразца, если он был получен из архива нуклеотидов (SRA). | Строго рекомендуется |
| samplingProtocol | UV light trap | Название, ссылка на или описание метода или протокола, использованного для отбора проб. https://dwc.tdwg.org/terms/#dwc:samplingProtocol | Рекомендуется |
| associatedSequences | https://www.ncbi.nlm.nih.gov/nuccore/MK405371 | Список (через ( | ) идентификаторов (публикация, глобальный уникальный идентификатор, URI) генетической последовательности, связанной со находкой (сиквенсом). Может использоваться для связи с архивами трейс-файлов сиквенсов. | Рекомендуется |
| identificationRemarks | RDP annotation confidence (at lowest specified taxon): 0.96, against reference database: GTDB | Детали о процессе таксономической идентификации, в идеале включая данные о примененном алгоритме и справочной базе данных, а также об уровне достоверности полученной идентификации. | Рекомендуется |
| identificationReferences | https://www.ebi.ac.uk/metagenomics/pipelines/4.1 https://github.com/terrimporter/CO1Classifier | Список ссылок на источник, использованный для идентификации таксонов (названия, DOI или URI). Названия разделяются ( | ) чертой. | Рекомендуется |
| decimalLatitude | 60.545207 | Географическая широта | Строго рекомендуется |
| decimalLongitude | 24.174556 | Географическая долгота | Строго рекомендуется |
| taxonID | ASV:7bdb57487bee022ba30c03c3e7ca50e1 | For eDNA data, it is recommended to use an MD5 hash of the sequence and prepend it with “ASV:”. See also §1.6. | Строго рекомендуется, if DNA_sequence is not provided |
| scientificName | Gadus morhua L. 1758, BOLD:ACF1143 | Полное научное название (вид или высший таксон) или идентификатор OTU из порталов BOLD (BIN) или UNITE (SH) | Обязательно |
| kingdom | Animalia | Царство | Строго рекомендуется |
| phylum | Chordata | Фила | Рекомендуется |
| class | Actinopterygii | Класс | Рекомендуется |
| order | Gadiformes | Порядок | Рекомендуется |
| family | Gadidae | Семейство | Рекомендуется |
| genus | Gadus | Род | Рекомендуется |
Рекомендуемые поля данных и метаданных о последовательностях (DNA-derived data)
| Название поля | Примеры | Описание | Обязательность |
|---|---|---|---|
| DNA_sequence | TCTATCCTCAATTATAGGTCATAATTCACCATCAGTAGATTTAGGAATTTTCTCTATTCATATTGCAGGTGTATCATCAATTATAGGATCAATTAATTTTATTGTAACAATTTTAAATATACATACAAAAACTCATTCATTAAACTTTTTACCATTATTTTCATGATCAGTTCTAGTTACAGCAATTCTCCTTTTATTATCATTA | Последовательность видовой гипотезы (OTU, ASV). | Строго рекомендуется |
| sop | https://www.protocols.io/view/emp-its-illumina-amplicon-protocol-pa7dihn | Стандартные операционные процедуры или пайплайны, использованные для секвенирования и обработки полученных сиквенсов. Может быть ссылкой на протокол, размещенный на портале protocols.io | Рекомендуется |
| target_gene | 16S rRNA, 18S rRNA, ITS | Секвенируемый ген (регион) | Строго рекомендуется |
| target_subfragment | V6, V9, ITS2 | Название фрагмента гена или маркера. Важно для идентификации специальных областей на маркерных генах, таких как гипервариабельная область V6 гена 16S рРНК | Строго рекомендуется |
| pcr_primer_forward | GGACTACHVGGGTWTCTAAT | Последовательность прямого праймера | Строго рекомендуется |
| pcr_primer_reverse | GGACTACHVGGGTWTCTAAT | Последовательность обратного праймера | Строго рекомендуется |
| pcr_primer_name_forward | jgLCO1490 | Название прямого праймера | Строго рекомендуется |
| pcr_primer_name_reverse | jgHCO2198 | Название обратного праймера | Строго рекомендуется |
| pcr_primer_reference | https://doi.org/10.1186/1742-9994-10-34 | Ссылка (цитата) праймера | Строго рекомендуется |
| env_broad_scale | forest biome [ENVO:01000174] | Классификатор биомов, высшая категория, рекомендуется пользоваться порталом ENVO: http://purl.obolibrary.org/obo/ENVO_00000428 | Рекомендуется |
| env_local_scale | litter layer [ENVO:01000338] | Классификатор биомов, локальные условия, рекомендуется пользоваться порталом ENVO: http://purl.obolibrary.org/obo/ENVO_00000428 | Рекомендуется |
| env_medium | soil[ENVO:00001998] | Классификатор биомов, субстрат, рекомендуется пользоваться порталом ENVO: http://purl.obolibrary.org/obo/ENVO_00000428 | Рекомендуется |
| lib_layout | Paired | Одно- или двустороннее прочтение | Рекомендуется |
| seq_meth | Illumina HiSeq 1500 | Метод и платформа секвенирования | Строго рекомендуется |
| otu_class_appr | «dada2; 1.14.0; ASV» | Алгоритм и параметры кластеризации для получения OTU и ASV | Строго рекомендуется |
| otu_seq_comp_appr | «blastn;2.6.0+;e-value cutoff: 0.001» | Интрумент для привязки видовых эпитетов к видовым гипотезам OTUs и ASVs | Строго рекомендуется |
| otu_db | «Genbank nr;221», «UNITE;8.2» | Ссылка на базу данных использованных для классификации | Строго рекомендуется |
